10月9日,中山大学医学院施莽教授团队与阿里云李兆融团队在《细胞》杂志(Cell)发表论文,报告了全球范围的180个超群、16万余种的RNA病毒发现,大幅扩展全球RNA病毒的多样性。该研究将人工智能技术应用于病毒鉴定,发现了传统研究方法未能发现的病毒“暗物质”,探索了病毒学研究的新路径。
传统的病毒发现方法包括病毒分离和生命组学的生物信息学分析,高度依赖既有知识,面对RNA病毒这种高度分化、种类繁多且容易变异的病毒识别效率低。在该研究中,团队开发的LucaProt人工智能算法能够对病毒和非病毒基因组序列深度学习,并在数据集中自主判断病毒序列。利用这套算法,研究团队在来自全球生物环境样本的10,487份RNA测序数据中发现了超过51万条病毒基因组,代表超过16万个潜在病毒种及180个RNA病毒超群。其中23个超群无法通过序列同源方法识别,被称为病毒圈的“暗物质”。
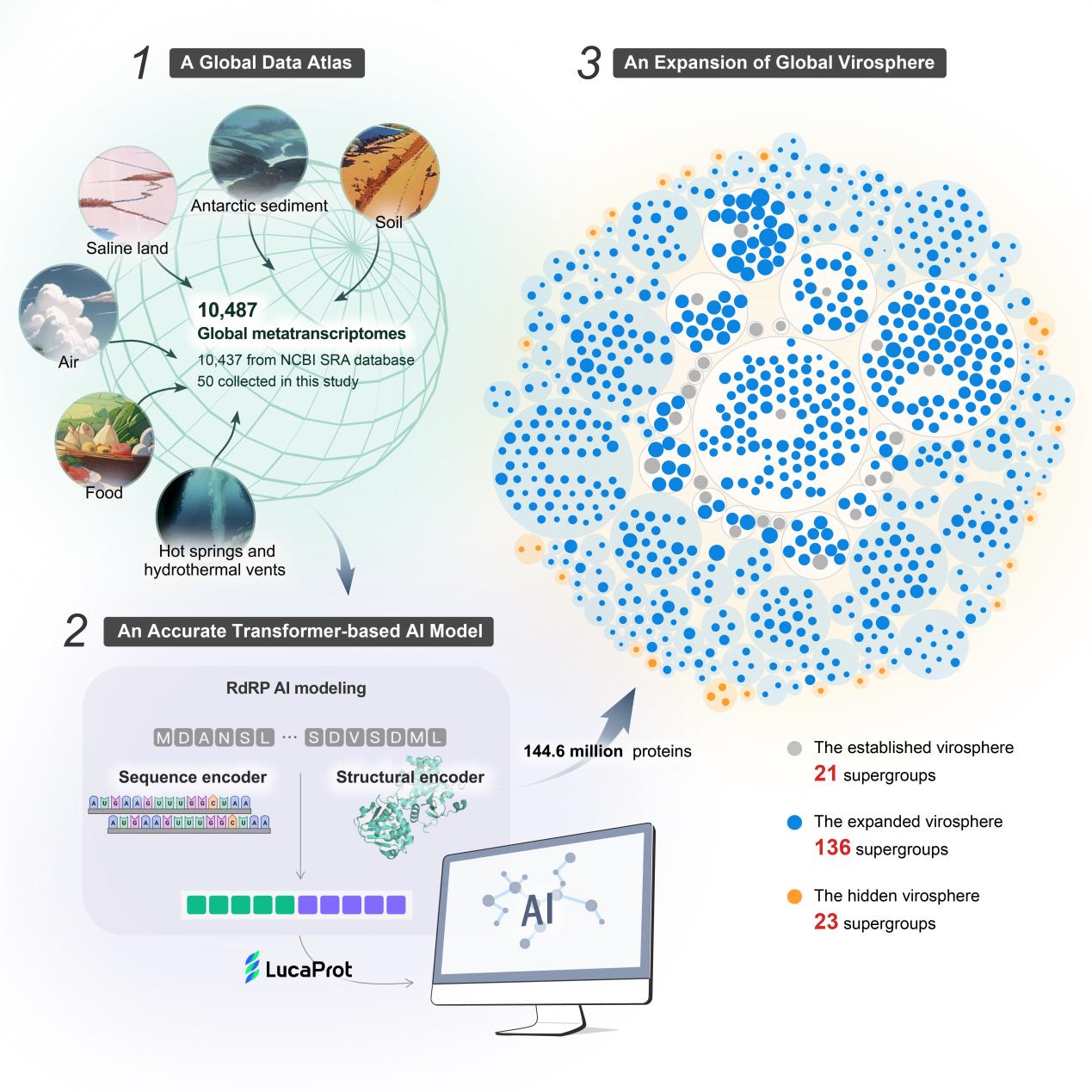
“人工智能的算法模型能够挖掘出我们之前忽略或根本不知道的病毒,这种能力在疾病防控和新病原的快速识别中尤为重要。特别是在疫情暴发时,人工智能的速度和精度可以帮助科学家更快地锁定潜在病原体。”施莽说。

通过进一步分析,团队报告了迄今最长的RNA病毒基因组,长度达到47,250个核苷酸;发现了超出以往认知的基因组结构,展现出RNA病毒基因组进化的灵活性;识别到多种病毒功能蛋白,特别是与细菌相关的功能蛋白,进一步表明还有更多类型的RNA噬菌体亟待探索;发现在南极底泥、深海热泉、活性污泥和盐碱滩等极端环境中,RNA病毒的数量和多样性仍然较高。新病毒的发现,刷新着科学家对病毒圈的认识。

“面对远源的新病毒,现有的病毒分类体系已经显得力不从心。未来,这一体系在门、纲等更深层次的分类上,可能会有大规模的调整。”施莽说,“我们的研究展示了病毒多样性的深度,但广度仍有待更多样本的补充。病毒的多样性远超人类想象,我们目前所看到的仍是冰山一角。”
施莽表示,这项研究与阿里云飞天实验室的AI4S-生物计算团队合作开展,希望未来继续通过跨领域科研合作,充分利用云计算和人工智能的优势,解决生命科学领域的重要问题。
论文链接:https://www.cell.com/cell/fulltext/S0092-8674(24)01085-7
文丨记者 陈亮
通讯员朱嘉豪 李建平
图丨学校供图